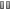| 29 Juin 2013
Le génome du virus PgV-16T, qui infecte une microalgue photosynthétique très commune, vient d’être décrypté par des chercheurs français, hollandais et américains, coordonnés par le laboratoire Information génomique et structurale (CNRS/Aix-Marseille Université). Ce nouveau virus présente des caractéristiques proches des virus géants de la famille des Megaviridae. Il est notamment la cible d’une infection virale persistante par un virophage, virus typique des Megaviridae, qui pourrait constituer un vecteur idéal pour l’échange de gènes entre virus et eucaryotes. Ces travaux ont été publiés dans la revue PNAS.
Après la découverte et le décryptage, il y a déjà dix ans, du génome du premier virus géant, baptisé « Mimivirus », la fouille bioinformatique des bases de données de séquences d'ADN isolées à partir d'environnements marins très variés a rapidement suggéré la présence d’une abondance de cousins de Mimivirus dans la mer. Les virus les plus grands, regroupés dans la famille des Megaviridae, ont tous été isolés dans des amibes du genre Acanthamoeba qui, bien qu'ubiquitaires, sont très peu représentées dans les océans, dominés par des espèces planctoniques photosynthétiques. La difficulté à isoler de nouveaux virus géants réside dans le fait que les organismes cellulaires victimes de leur infection sont encore méconnus.
Dans ce contexte, un progrès décisif vient d'être accompli avec le décryptage du génome du virus PgV-16T, qui infecte une microalgue unicellulaire très commune, Phaeocystis globosa. Cette algue, qui appartient à l’embranchement des Haptophytes, domine la communauté phytoplanctonique sur les côtes de l'Atlantique et de la Mer du Nord à l'occasion d'efflorescences spectaculaires produisant d'importantes quantités de mousse blanchâtre sur les plages. Le virus PgV-16T participe à l'interruption de ces efflorescences et constitue donc un acteur majeur de l'équilibre écologique des différentes populations planctoniques.
Le génome du virus PgV-16T est une molécule d'ADN constituée de 460 000 paires de nucléotides, qui code pour 434 protéines. Bien que ne possédant que la moitié du millier de gènes que comptent les Megaviridae, ce virus se réplique par un mécanisme très similaire. Chaque particule virale de PgV-16T renferme également plusieurs molécules d'ADN beaucoup plus petites, d’environ 20 000 paires de nucléotides. La séquence de ces petites molécules d’ADN est similaire à celle des « virophages », virus trouvés exclusivement en association avec les virus de la famille des Megaviridae. Parmi les 16 protéines codées par cet ADN accessoire, aucune ne ressemble à une protéine de capside absolument nécessaire à la formation de particules virales infectieuses. Ce petit génome est donc celui d'un virophage défectif ayant perdu son propre véhicule et qui ne peut se transmettre qu'en empruntant la particule de son hôte, le virus PgV-16T. Cette absence de véhicule propre fait de ce virophage l'équivalent des plasmides hébergés par les bactéries et en dénote peut-être l'origine évolutive commune. Comme les plasmides, qui sont les vecteurs principaux de l'échange des gènes dans le monde bactérien, les virophages pourraient donc être des vecteurs importants de l'échange de gènes entre organismes eucaryotes.
Ces travaux montrent donc qu’une fraction significative du plancton photosynthétique est bien la cible de virus géants. Les Megaviridae, dont l’origineévolutive est contemporaine de celle des eucaryotes, sont donc depuis toujours des régulateurs des populations planctoniques à la base des réseaux trophiques océaniques, grandes consommatrices de CO2 atmosphérique et productrices de la moitié de l'oxygène de notre planète.

Figure : Particules du virus PgV-16T (hexagones sombres) au sein d’une cellule de Phaeocystis globosa observée en microscopie électronique. © IGS, CNRS/Aix-Marseille Université