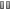| 26 Mai 2011
 Des chercheurs du CEA, du CNRS et de l’Université Joseph Fourier (IBS[1], UVHCI[2], AFMB[3]) viennent d’observer pour la première fois la partie désordonnée de la nucléoprotéine du virus de la rougeole dans un contexte physiologique. En utilisant une combinaison de trois techniques complémentaires[4], la microscopie électronique (EM), la diffusion des rayons X aux petits angles (SAXS) et la résonance magnétique nucléaire (RMN) en solution, ce consortium a pu proposer un modèle intégral de la nucléocapside[5] qui contient l’ARN du virus. Leurs résultats suggèrent que cette partie désordonnée joue un rôle dans l’infection par le virus de la rougeole. Ces travaux sont publiés le 26 mai sur le site de la revue PNAS.
Des chercheurs du CEA, du CNRS et de l’Université Joseph Fourier (IBS[1], UVHCI[2], AFMB[3]) viennent d’observer pour la première fois la partie désordonnée de la nucléoprotéine du virus de la rougeole dans un contexte physiologique. En utilisant une combinaison de trois techniques complémentaires[4], la microscopie électronique (EM), la diffusion des rayons X aux petits angles (SAXS) et la résonance magnétique nucléaire (RMN) en solution, ce consortium a pu proposer un modèle intégral de la nucléocapside[5] qui contient l’ARN du virus. Leurs résultats suggèrent que cette partie désordonnée joue un rôle dans l’infection par le virus de la rougeole. Ces travaux sont publiés le 26 mai sur le site de la revue PNAS.
Le postulat central qui a mondialement motivé les investissements dans des projets de biologie structurale est fondé sur l'hypothèse que la détermination de la structure tridimensionnelle d'un nombre fini de protéines fournira la clef pour comprendre leurs activités biologiques. Cependant, au cours de la dernière décennie, il est devenu de plus en plus clair qu'une fraction importante (jusqu'à 40%) des protéines codées par le génome humain sont intrinsèquement désordonnées ou contiennent des régions désordonnées de longueur importante (> 50 acides aminés). Les protéines intrinsèquement désordonnées (PIDs) restent fonctionnelles malgré l'absence d'une structure tridimensionnelle bien définie. Le paradigme « structure-fonction » classique ne s’applique donc pas à ces protéines et de nouvelles études sur la relation entre séquence primaire et fonction moléculaire sont nécessaires, ainsi que de nouvelles méthodes pour observer ces parties flexibles des protéines.
C’est dans ce contexte et dans le cadre du « Partenariat pour la biologie structurale » de Grenoble[6] que les chercheurs du CEA, du CNRS et de l’Université Joseph Fourier se sont intéressés à la nucléoprotéine du virus de la rougeole, dont la nature partiellement désordonnée contrôle la réplication du virus. Cette protéine forme des nucléocapsides en se liant avec l’ARN viral (voir figure). Les chercheurs ont mis au point un modèle qui reconstruit la structure de la nucléocapside à partir des données expérimentales obtenues par trois techniques complémentaires : l’EM, la SAXS et la RMN. Cette étude révèle pour la première fois le domaine désordonné dans le contexte de la nucléocapside entière (in situ).
Les résultats montrent que cette partie de la protéine, qui contrôle la transcription et la réplication du virus, reste bien désordonnée in situ. De plus, ces résultats suggèrent très fortement que la flexibilité intrinsèque de la protéine joue un rôle essentiel dans sa fonction. Avec ces techniques, les chercheurs vont désormais pouvoir étudier la structure, la dynamique et la cinétique de la nucléoprotéine in situ, en présence de l’ARN polymérase[7] pour aller plus loin dans la compréhension des mécanismes conduisant à la propagation du virus.
Figure : la résonance magnétique nucléaire (spectre en haut à gauche) et la microscopie électronique (au milieu à gauche) ont été combinées avec la technique de diffusion aux petits angles (en bas à gauche), pour développer un modèle de la nucléocapside entière du virus de la rougeole (à droite). Les domaines structurés de la nucléoprotéine (verts et jaunes) lient l’ARN (bleu) tandis que le domaine désordonné (rouge) qui contrôle la transcription et la réplication du virus, sort de l’espace de la capside pour interagir avec l’ARN polymérase. Au centre : un détail de la structure de la nucléocapside (en haut) et la structure d’un tour complet d’hélice (en bas).
------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
Référence de l’article :
Intrinsic disorder in measles virus nucleocapsids. Malene Ringkjøbing Jensen, Guillaume Communie, Euripedes Almeida Ribeiro Jr, Nicolas Martinez, Ambroise Desfosses, Loïc Salmon, Luca Mollica, Frank Gabel, Marc Jamin, Sonia Longhi, Rob W. H. Ruigrok, and Martin Blackledge. PNAS, online, 2011.
------------------------------------------------------------------------------------------------------------------------------------------------------------------------------
Contact presse :
CEA : Coline Verneau – 01 64 50 14 88 –
Cette adresse email est protégée contre les robots des spammeurs, vous devez activer Javascript pour la voir.
-----------------------------------------------------------------------------------------------------------------------------------------------------------------------------
[1] Institut de biologie structurale Jean-Pierre Ebel (IBS, Institut mixte CEA-CNRS-Université Joseph Fourier, Grenoble)
[2] Unit of Virus Host Cell Interactions (UVHCI) UMI 3265 UJF-EMBL-CNRS Grenoble
[3] Architecture et Fonction des Macromolécules Biologiques UMR 6098 CNRS et Universités d'Aix-Marseille I et II
[4] La microscopie électronique donne une image globale de la partie structurée de ces gros objets ; la technique dite de diffusion aux petits angles mesure le diamètre de la capside et permet donc de voir dans quel périmètre se produisent les mouvements des parties flexibles des protéines ; la RMN (résonance magnétique nucléaire) détermine la dynamique de l’ensemble du domaine désordonné de la protéine avec une résolution atomique.
[5] Nucléocapside du virus : structure complexe constituée d’un ARN en hélice associé à 13 molécules de nucléoprotéine par tour d’hélice.
[6] Partenariat pour la biologie structurale : alliance constituée par les acteurs grenoblois de la biologie structurale, l’IBS, l’EMBL, l’ESRF et l’ILL.
[7] L’ARN polymérase est l’enzyme en charge de la réplication de l’ARN du virus, étape indispensable à sa propagation. Elle interagit directement avec la nucléoprotéine.